AlphaFolds nouveau rival ? Meta AI prédit la forme de 600 millions de protéines
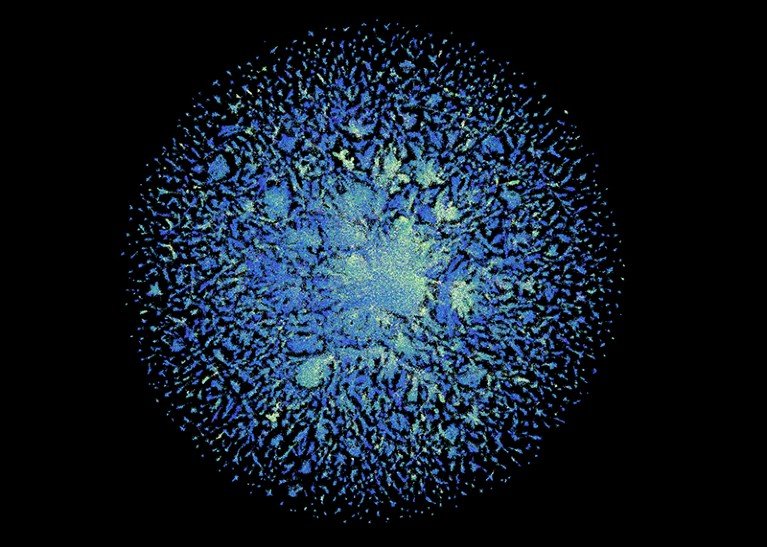
L’Atlas métagénomique ESM contient des prédictions structurelles pour 617 millions de protéines.Crédit : Atlas métagénomique ESM (CC BY 4.0)
Lorsque la société londonienne d’intelligence artificielle (IA) DeepMind a dévoilé les structures prédites de quelque 220 millions de protéines plus tôt cette année, le trésor couvrait presque toutes les protéines d’organismes connus dans les bases de données ADN. Maintenant, un autre géant de la technologie remplit la matière noire de l’univers des protéines.
Les chercheurs de Meta (anciennement Facebook, dont le siège est à Menlo Park, Californie) ont utilisé l’IA pour prédire les structures de quelque 600 millions de protéines de bactéries, virus et autres micro-organismes qui n’ont pas été caractérisés.
Cela va tout changer : DeepMinds AI fait un pas de géant dans la résolution des structures protéiques
Ce sont les structures que nous connaissons le moins. Ce sont des protéines incroyablement mystérieuses. Je pense qu’ils offrent le potentiel d’une grande compréhension de la biologie, déclare Alexander Rives, responsable de la recherche de l’équipe des protéines Meta AIs.
Les scientifiques ont généré les prédictions décrites dans une préimpression du 1er novembre1 en utilisant un grand modèle de langage, un type d’IA qui peut prédire le texte à partir de quelques lettres ou mots seulement.
Normalement, les modèles de langage sont entraînés sur de gros volumes de texte. Pour les appliquer aux protéines, Rives et ses collègues ont plutôt alimenté les séquences d’IA de protéines connues, qui peuvent être écrites sous la forme d’une série de lettres, chacune représentant l’un des 20 acides aminés possibles. Le réseau a alors appris à remplir les séquences de protéines dans lesquelles certains des acides aminés étaient obscurcis.
Autocomplétion des protéines
Cette formation a imprégné le réseau d’une compréhension intuitive des séquences de protéines, qui contiennent des informations sur leurs formes, explique Rives. Une deuxième étape inspirée par l’IA pionnière de DeepMinds en matière de prédiction de la structure des protéines, AlphaFold combine ces informations avec des informations sur les relations entre les structures et les séquences protéiques connues, pour générer des prédictions.
Le réseau Metas, appelé ESMFold, n’est pas aussi précis qu’AlphaFold, a rapporté l’équipe Rives plus tôt cette année2, mais il est environ 60 fois plus rapide pour prédire les structures de séquences courtes, dit-il. Cela signifie que nous pouvons adapter la prédiction de la structure à des bases de données beaucoup plus grandes.
À titre de test, les chercheurs ont lancé leur modèle sur une base de données d’ADN métagénomique séquencé en masse provenant de sources environnementales telles que le sol, l’eau de mer et l’intestin et la peau humains. La grande majorité des entrées qui codent pour des protéines potentielles proviennent d’organismes unicellulaires qui n’ont jamais été isolés ou cultivés et qui sont inconnus de la science.
Au total, l’équipe a prédit les structures de plus de 617 millions de protéines. L’effort n’a pris que deux semaines (en revanche, AlphaFold peut prendre quelques minutes pour générer une seule prédiction). Les structures sont disponibles gratuitement, tout comme le code sous-jacent au modèle, explique Rives.

Quelle est la prochaine étape pour AlphaFold et la révolution du repliement des protéines de l’IA
Sur les 617 millions de prédictions, le modèle a estimé que plus d’un tiers étaient de haute qualité, de sorte que les chercheurs peuvent être sûrs que la forme globale de la protéine est correcte et, dans certains cas, peuvent discerner des détails au niveau atomique. Des millions de ces structures ne ressemblent absolument à rien dans les bases de données de structures protéiques déterminées expérimentalement, ni à aucune des prédictions AlphaFolds d’organismes connus.
Une grande partie de la base de données AlphaFold est composée de structures presque identiques les unes aux autres, alors que les bases de données métagénomiques devraient couvrir une grande partie de l’univers protéique jamais vu auparavant, explique Martin Steinegger, biologiste informatique à l’Université nationale de Séoul. Il y a maintenant une grande opportunité de démêler davantage les ténèbres.
Sergey Ovchinnikov, biologiste de l’évolution à l’Université de Harvard à Cambridge, Massachusetts, s’interroge sur les centaines de millions de prédictions qu’ESMFold a faites avec une faible confiance. Certains peuvent ne pas avoir de structure définie, du moins de manière isolée, tandis que d’autres peuvent être de l’ADN non codant confondu avec du matériel codant pour des protéines. Il semble qu’il y ait encore plus de la moitié de l’espace protéique dont nous ne savons rien, dit-il.
Plus léger, plus simple, moins cher
Burkhard Rost, biologiste informatique à l’Université technique de Munich en Allemagne, est impressionné par la vitesse et la précision combinées du modèle Metas. Mais il se demande si ESMFold offre vraiment un avantage sur la précision d’AlphaFolds lorsqu’il s’agit de prédire les protéines à partir de bases de données métagénomiques. Méthodes de prédiction basées sur un modèle de langage, dont une développée par son équipe3 sont mieux adaptés pour déterminer rapidement comment les mutations modifient la structure d’une protéine, ce qui n’est pas possible avec AlphaFold. Nous verrons la prédiction de la structure devenir plus légère, plus simple, moins chère, et cela ouvrira la porte à de nouvelles choses, dit-il.
DeepMind n’a actuellement pas l’intention d’inclure des prédictions structurelles métagénomiques dans sa base de données, mais n’a pas exclu de les ajouter aux futures versions, selon un représentant de l’entreprise. Mais Steinegger et ses collaborateurs ont utilisé une version d’AlphaFold pour prédire les structures de quelque 30 millions de protéines métagénomiques. Ils espèrent trouver de nouveaux types de virus à ARN en recherchant des formes jusque-là inconnues des enzymes copiant le génome du virus.
Steinegger voit la matière noire des biologies de chalutage comme la prochaine étape évidente pour de tels outils. Je pense que nous aurons bientôt une explosion dans l’analyse de ces structures métagénomiques.